识别干扰世界各大洲人群遗传结构推断的基因组区域
- 2022-11-23 00:00
- 来源:医药资讯网
- 阅读:233
来源:互联网2022-11-23 19:24
自从人类的祖先离开非洲以来,迁徙、基因混合、自然选择等诸多历史事件都在基因组上留下了印记,形成了群体遗传结构。
自从人类的祖先离开非洲以来,迁徙、基因混合、自然选择等诸多历史事件都在基因组上留下了印记,形成了群体遗传结构。因此,推断群体的遗传结构对于研究群体的进化史非常重要,也有助于纠正全基因组关联研究中群体结构的混杂效应()。2008年,Price等人发现欧洲人群基因组中的某些区域会干扰人群遗传结构的推断。然而,不同大陆的人有不同的进化史,而且近年来基因检测技术和人群基因组数据日新月异,迫切需要对这一问题进行系统的分析和更新。
近日,华中科技大学公共卫生学院王教授团队基于千人基因组计划38版人类参考基因组的高深度(30)全基因组测序数据,通过系统分析,在欧洲、东亚、南亚和非洲发现了干扰人群遗传结构推断的27个区域。当将主成分分析应用于群体,或者用混合软件推断群体的遗传结构时,排除这27个基因组区域可以得到更准确的推断结果。相关文章(点击文末阅读原文,下载完整PDF)发表在《定量生物学》杂志上。

全文摘要
在本文中,作者将对推断群体遗传结构做出异常贡献的基因组区域定义为异常区域。作者创建了一套识别这些异常区域的生物信息学分析流程,并将其应用于四大洲的人口数据。
首先,通过PCA从质量控制后的基因型数据中推断群体的遗传结构,并获得每个突变位点对推断结果的影响,据此确定对推断结果产生异常影响的基因组区域。去除这些区域后,基于剩余区域的基因型再次进行PCA。这个过程重复迭代多次,直到下一轮PCA没有新的异常区域出现。我们结合每次迭代中发现的区域,这是这个大陆上的人的最终不寻常的区域(见图1)。作者在欧洲人群中发现了17个这样的区域,东亚13个,南亚10个,非洲5个。随着各大洲内遗传多样性的增加,这种区域的数量减少。
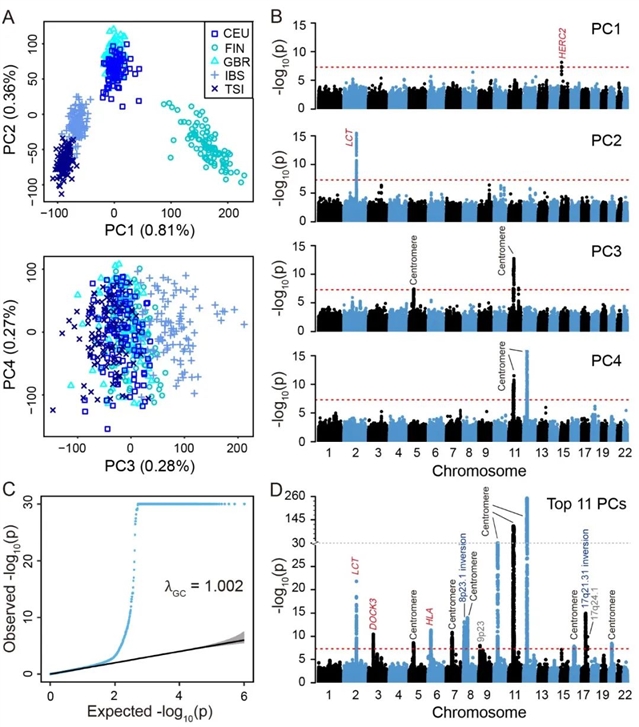
图一。欧洲人口中不寻常的区域
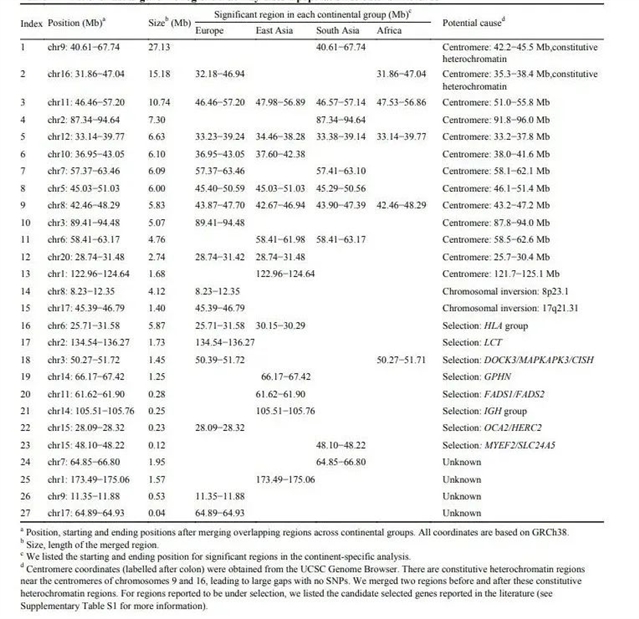
根据可能的原因,作者将27个异常区域分为四类:位于染色体着丝粒附近的区域(13),经历染色体倒位的区域(2),受到自然选择的区域(8)和未知原因的区域(4)(见表1)。中心粒附近区域在正常细胞分裂中起重要作用,其重组率很低,导致一个长的连锁不平衡(LD)区。此外,染色体倒位和自然选择会导致强烈的LD。
表1。影响群体遗传结构推断的基因组异常区域
然后通过对比去除异常区域前后的混合分析结果,作者发现欧洲人群的群体遗传结构推断受影响最大,去除后的结果更符合欧洲人的进化史(见图2)。也说明在遗传多样性较低的群体中,这些区域对群体遗传结构推断结果的影响更为明显,与相关研究结果一致。
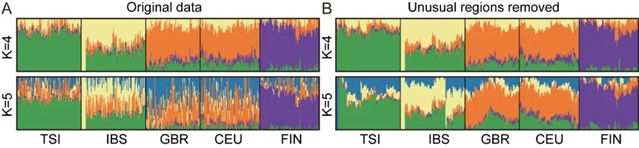
图二。欧洲人群中异常区域对混合结果的影响
最后,作者建议在利用人类基因型数据推断群体遗传结构时,应首先排除本研究确定的27个异常区域的遗传变异位点,以获得对群体遗传结构的准确推断,有利于下游的进化史分析或全基因组关联研究。
版权声明
本网站所有标注“来源:100医学网”或“来源:bioon”的文字、图片及音视频资料,版权归100医学网网站所有。未经授权,任何媒体、网站、个人不得转载,否则将追究法律责任。获得书面授权转载时,必须注明“来源:100医学网”。其他来源的文章均为转载文章。本网站所有转载文章都是为了传递更多信息。转载内容不代表本站立场。不想被转载的媒体或个人可以联系我们,我们会立即删除。
87%的用户都在使用100医疗网APP随时阅读、评论、分享、交流。请扫描二维码下载-

