《自然》子课题:陆地团队合作开发基于人工智能技术的抗体研究模型
- 2022-11-14 00:00
- 来源:医药资讯网
- 阅读:36
来源:生物世界2022-11-14 09336038
在本研究中,首次提出了深度Ab-Ag相互作用算法(简称DeepAAI)。DeepAAI不同于经典的序列比对方法,而是通过深度学习。
在人类与病毒病原体的博弈中,发现强大的中和抗体(nab)是治疗中要使用的重要武器之一。在天然抗体或人工设计的抗体中,作用机制和中和能力的研究过程往往需要大量的实验来检测和探索,也是人类与病毒赛跑中的关键限速环节。如何快速预测未知抗体的中和能力和靶点,是传统抗体药物研发领域仍需进一步突破的关键科学问题。
复旦大学基础医学院的land团队与sensetime的张少婷和张捷一起发表了一篇题为《通过自然机器智能中的自适应图神经网络预测未知抗体的中性》的研究论文。
本研究首次提出了深度抗体-抗原相互作用算法(简称DeepAAI)模型。DeepAAI不同于经典的序列比对方法,而是通过深度学习动态、自适应地学习未知抗体与已知抗体的关系图,从而避免AI算法对未知抗体的冷启动,有效预测未知抗体的中和能力。此外,DeepAAI具有良好的解释特性,可以为抗原和抗体的结合位点提供线索。分析同一病毒的不同变种和亚变种之间的相似性,推荐一个病毒新亚种可能的中和抗体。

DeepAAI的另一个特点是基于序列数据。虽然真实的三级结构数据可以提高AI算法的预测精度,但是现实世界中大量抗体的三级结构是未知的。AAI抛弃了串联AI算法的模型,先根据序列预测结构,再根据预测的结构预测抗原抗体相互作用。而是直接基于序列提取足够多的有效特征来预测交互。这避免了步骤1中的误差在步骤2中被累积和指数放大的风险。同时,现实世界中大量的序列数据也可以增强AI算法的实用性。
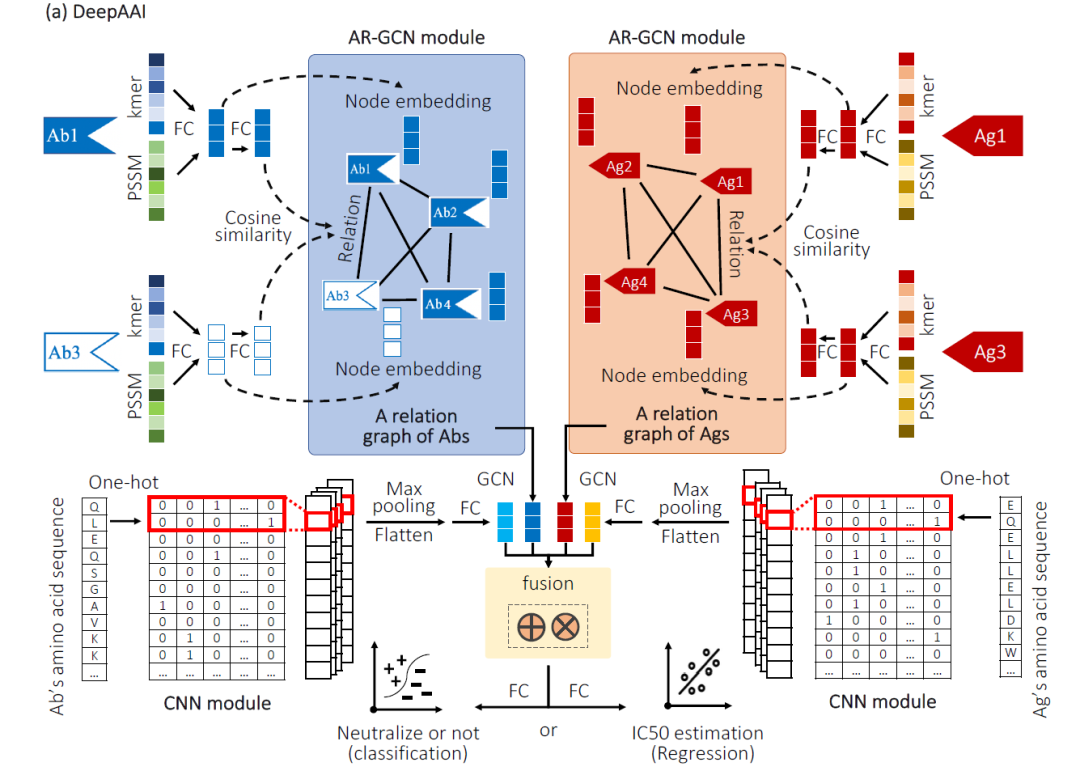
为了全面评估DeepAAI的预测能力,本研究以HIV、新冠肺炎(新型冠状病毒)、流感病毒(influenza)和登革热病毒为模型病毒,进行了深入的研究。DeepAAI对这些病毒的抗体中和能力显示了一定程度的准确预测。
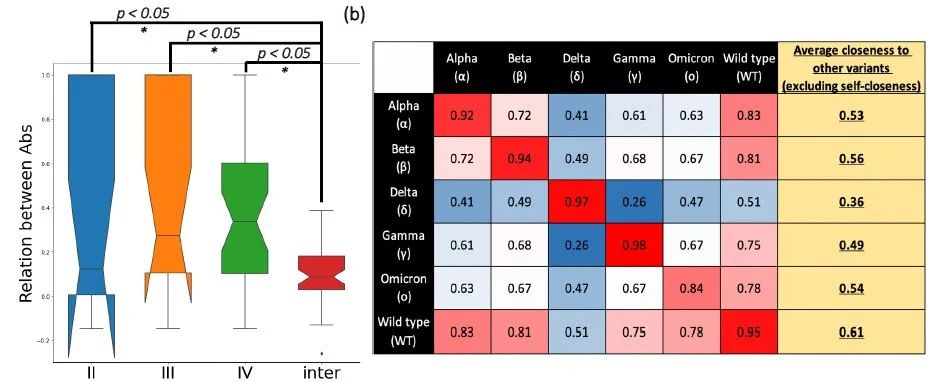
考虑到大量新型冠状病毒变异体的出现,尤其是Omicron亚型,该研究所报道的DeepAAI模型可能为抗体药物的优化和广谱抗病毒抗体的研发提供思路。
版权声明
本网站所有标注“来源:100医学网”或“来源:bioon”的文字、图片及音视频资料,版权归100医学网网站所有。未经授权,任何媒体、网站、个人不得转载,否则将追究法律责任。获得书面授权转载时,必须注明“来源:100医学网”。其他来源的文章均为转载文章。本网站所有转载文章都是为了传递更多信息。转载内容不代表本站立场。不想被转载的媒体或个人可以联系我们,我们会立即删除。
87%的用户都在使用100医疗网APP随时阅读、评论、分享、交流。请扫描二维码下载-

